動物遺伝制御学研究室
Laboratory of Animal Genetics
Laboratory of Animal Genetics
脊椎動物の比較ゲノム学(Comparative Genomics)
- 染色体・ゲノムの構造と機能から進化を探る -
- 染色体・ゲノムの構造と機能から進化を探る -
2.機能遺伝子の染色体マッピングによる核型進化の解析
3.脊椎動物にみられるゲノムのモザイク構造とその起源
さらに詳しい説明はこちら(pdfファイル) .
ある動物種から得られた染色体特異的DNAプローブを蛍光標識し、別種の染色体にハイブリダイズすることによって、異なる種間で相同な染色体領域を検出することができます。図1は、ニワトリとキジ科4種の間で生じた構造変化を染色体ペインティングプローブ法で検出した例を示しています。図2は、マウスの染色体DNAを用いた染色体ペインティングによって作成された、5種のネズミ亜科動物の染色体地図です。染色体上のブロックの番号は、対応するマウスの染色体番号を表しています。これらの結果を、系統樹の上にプロットすれば、図3に示すように、様々な動物の進化過程で生じた染色体構造変化のプロセスを推定することができます。さらに、系統樹を遡っていけば、既に絶滅した祖先がもっていた核型を推定することも可能です。最近の私たちの研究で、キジ目の祖先は、ダチョウやエミューなどのダチョウ目の鳥類がもつ核型と類似した核型をもっていたことがわかりました。また、ネズミ科動物の祖先核型も推定することができました。現在は、北海道大学理学研究員の西田千鶴子先生との共同研究で、さらに多くの鳥類について解析を行い、鳥類における核型進化過程の全貌を明らかにすることを試みています。
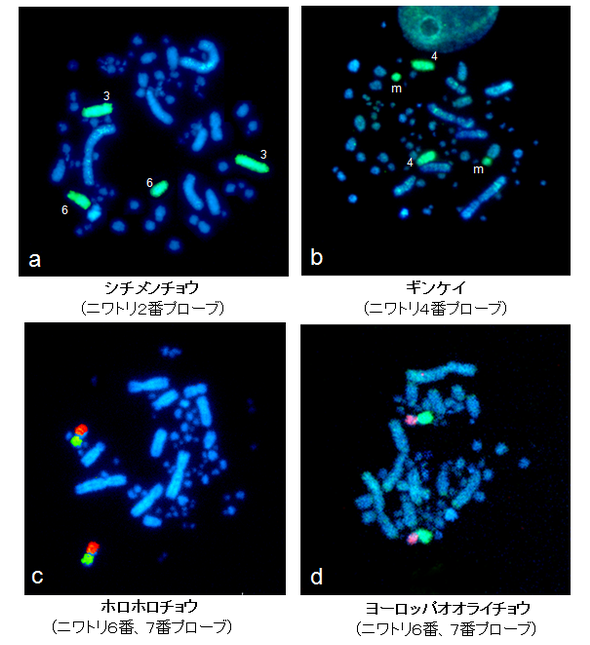
↑図1 ニワトリ染色体 DNA を用いたキジ科鳥類の染色体ペインティング
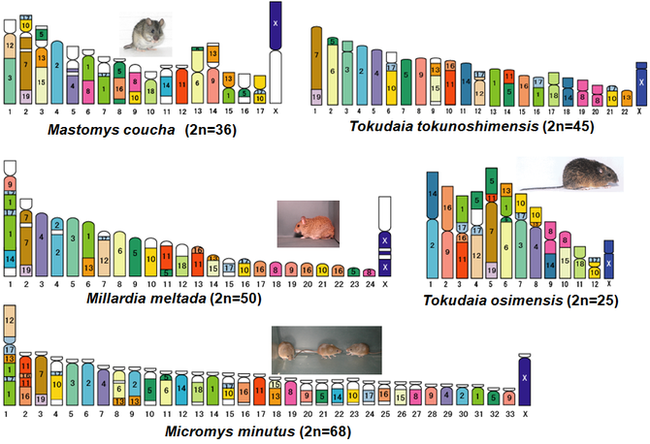
↑図2 ネズミ亜科 5種におけるマウスとの染色体相同領域の比較染色体地図
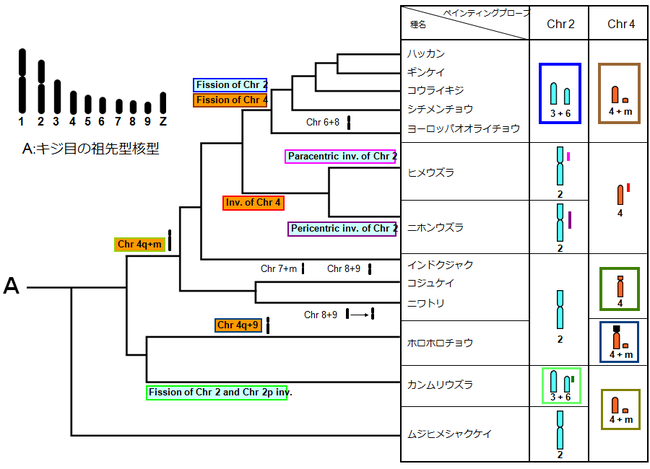
↑図3 ミトコンドリア Cytb 遺伝子の塩基配列から作成されたキジ目13種の分子系統樹と比較染色体ペインティングの結果から推定された、キジ目の核型進化の過程とキジ目の共通祖先の核型
鳥類や爬虫類の多くは、染色体サイズが大きく異なるマクロ染色体とマイクロ染色体で構成される特異的な核型をもっています(図4)。鳥類と爬虫類が約2億年以上前に共通祖先から分岐したことを考えると,鳥類と爬虫類間にみられる核型の類似性は、脊椎動物の核型進化と性染色体の進化の過程を考察する上で非常に興味ある問題を提起します.しかし、この核型の類似性が遺伝子の連鎖群の相同性を示すものか、あるいは収斂による偶然の事象なのかについては長い間不明でした.鳥類と爬虫類の染色体の相同性を調べる場合、両者は分類群が綱のレベルで異なり、染色体を構成するDNA配列も大きく異なるため、ニワトリのペインティングプローブを爬虫類の染色体解析に用いることはできません。しかし、脊椎動物が共通にもっている機能遺伝子を染色体上にマッピングすれば(図5)、哺乳類−鳥類−爬虫類間で直接に染色体の相同性を比較することが可能です。
私たちは、京都大学理学研究科の阿形清和博士との共同研究で、スッポンから得られた大量の遺伝子DNAクローンをスッポン染色体上にマッピングし、世界初の鳥類?爬虫類間の比較染色体地図の作成に成功しました。その結果、1-6番までのマクロ染色体では、ニワトリーカメ間で1対1の染色体の対応がみられ、ニワトリとカメは約2億3千年前に分岐したにもかかわらず、両者の染色体構造はほとんど変化していないことが明らかとなりました。その一例として、図6にニワトリとスッポンの1番染色体の比較地図を示しました。この図の真中にはヒト染色体との相同領域を表したニワトリの遺伝連鎖地図を示してあります。同様にシマヘビにおいても比較染色体地図を作製し、ニワトリの染色体地図と比較したところ、シマヘビではニワトリとの間にスッポンのような1対1の染色体の対応は見られず、ニワトリとの染色体相同性はカメより低いことが判明しました(図7)。これらの結果は、カメの方がヘビよりもより鳥類に近縁であるという分子系統学的な類縁関係を支持しています。私たちは、さらに多くの爬虫類動物について染色体地図を作成し、鳥類と爬虫類の間で、どのようなメカニズムで、そしてどのような過程を経て染色体進化が生じてきたかを明らかにしたいと考えています。また、現存する爬虫類の染色体の比較から、既に絶滅した恐竜や中生代の鳥類の染色体構造を推定するという大きな夢を実現したいと思っています。

↑図4 羊膜類の系統関係と核型
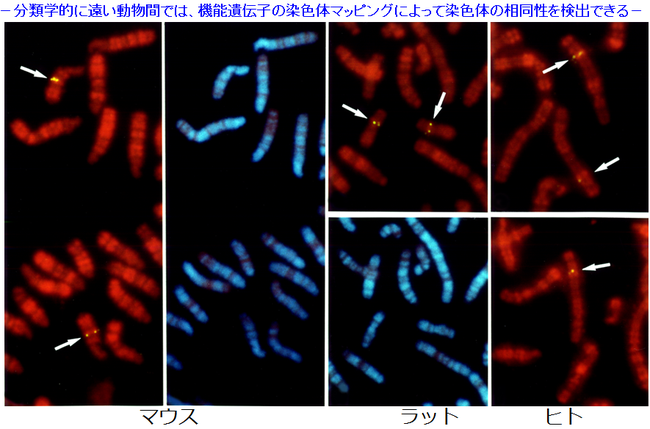
↑図5 ヒト、マウス、ラットにおける CNR8 遺伝子の比較染色体マッピング
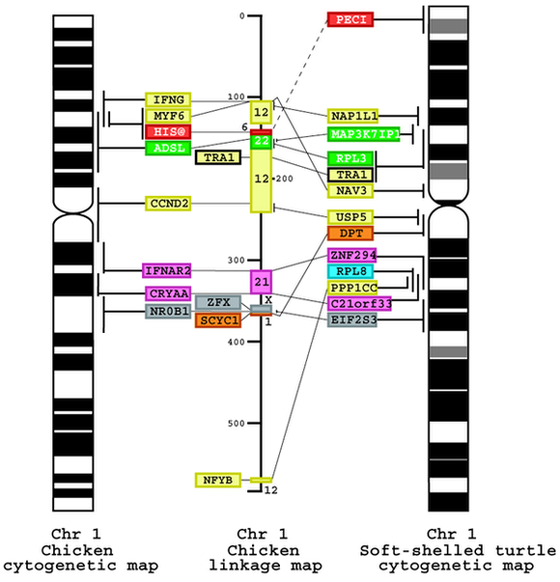
↑図6 Comparative cytogenetic map of chicken and turtle chromosome 1.
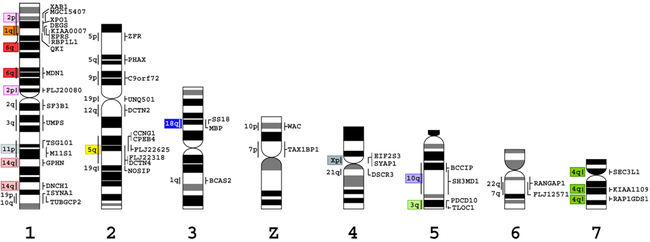
↑図7 シマヘビの機能遺伝子染色体地図(シマヘビのマクロ染色体にマップされた遺伝子のニワトリ染色体上の位置を、シマヘビ染色体の左側に示してあります。)
脊椎動物のゲノム中ではATGCの4文字が全くランダムに並んでいるのか? 鳥類と爬虫類がもつマイクロ染色体はどのような構造をしているのか? がよく判っていませんでした。ところが最近の研究で、哺乳類と鳥類のゲノムには、AT-rich(ATが豊富)な領域とGC?rich(GCが豊富)な領域が300 kb以上のサイズでモザイク状に存在すること、そして、ニワトリではマイクロ染色体はGC含量と遺伝子密度が高いことが明らかになってきました(図8)。しかし、このゲノムのモザイク構造が、変温動物(魚類、両生類、爬虫類)と恒温動物(鳥類、哺乳類)の分岐点で生じたという説と、羊膜をもたない動物(魚類、両生類)から羊膜をもつ動物(爬虫類、鳥類、哺乳類)に進化した時に生じたという説がありました。この謎を解明する鍵は、爬虫類が握っています。そこで私たちは、理化学研究所の工楽樹洋博士との共同研究で、スッポンのマクロ染色体とマイクロ染色体に存在する遺伝子間でGCの含量を比較し爬虫類ゲノム構造の特徴を調べました.その結果、ニワトリとスッポンでは、マイクロ染色体に存在する遺伝子群は、マクロ染色体の遺伝子群に比べて高いGC含量をもち、染色体のサイズに依存したGCの区画化、つまりマイクロ染色体にGCが有意に蓄積されていることを見つけました。この結果は、羊膜類の祖先では既にGC含量が区画化されたゲノム構造が獲得されていたことを示しています.そして、羊膜類におけるゲノム進化を考える場合、哺乳類では個々の染色体上でGC-richな領域とGC-poorな領域が同一染色体上でモザイク状に存在するのに対し、鳥類と爬虫類では、染色体のサイズ依存的なGC含量の区画化が生じている、つまりGC-richな部分はマイクロ染色体に集約されているという二つのモードが存在することを明らかにしました(図9)。そして、マウスなどの齧歯類の系統においては、ヒトに比べてGC含量の二次的な均一化が進んでいると考えられます.私たちは、マイクロ染色体をもつ他の爬虫類(ヘビ類)とマイクロ染色体をもたない爬虫類(ワニ)をもちいて、さらに詳細な解析を進めています。また、今後は、このようなゲノムのモザイク構造を産み出した要因として、核内の染色体テリトリーに着目し、クロマチンの核ダイナミクスと核型進化の関係について研究を行っていく予定です。
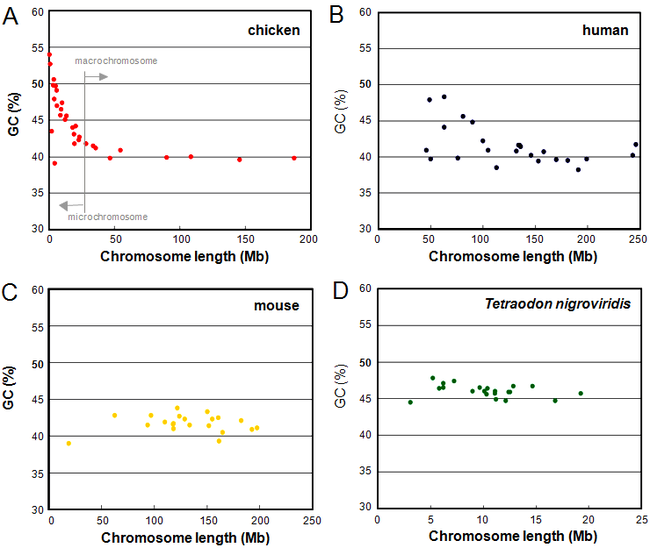
↑図8 染色体サイズとGC含量の関係(横軸が染色体サイズごとのGC含量、縦軸に各染色体DNA全体のGC含量を示す。)
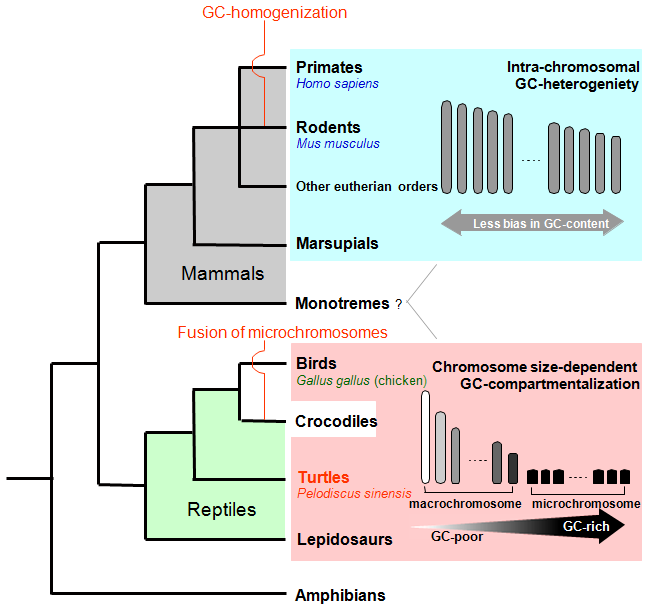
↑図9 Chromosomal evolution and GC heterogeneity in amniote phylogeny